Analyse de références bibliographiques avec R
Des graphiques et des nuages de mots sur les références bibliographiques rassemblées au cours de mon doctorat

Pendant mon doctorat, j’ai travaillé à l’intersection de la biophysique et du développement des plantes. Mon sujet de doctorat portait sur l’étude du rôle des contraintes mécaniques sur la division cellulaire. Pour explorer ce sujet, j’ai fait beaucoup de microscopie confocale, mais aussi beaucoup d’analyse d’images biologiques et d’analyse de données. J’étais curieuse de voir si ma bibliographie de thèse reflètait mon sujet. Pour analyser ces données, j’ai utilisé le logiciel R.
Une partie de mon travail de chercheur consiste à lire des articles de recherche pour élargir mes connaissances et me tenir au courant des progrès de la recherche dans mon domaine. J’utilise Zotero pour classer toutes les références aux articles que j’ai dans mon dossier de bibliographie Pendant mon doctorat, j’utilisais une extension de Zotero, Paper Machines, pour créer des nuages de mots. J’aimais bien visualiser les mots les plus fréquents présents dans ma bibliographie. Malheureusement, Paper Machines n’est plus maintenu. Cela m’a amenée à chercher comment créer des nuages de mots avec R, mais aussi comment explorer les données bibliographiques en général. Dans cet article, je rassemble mes trouvailles et propose quelques exemples de visualisation graphique des références bibliographiques rassemblées au cours de mon doctorat. Je me suis inspirée de deux autres articles de blog : https://davidtingle.com/misc/bib et https://www.littlemissdata.com/blog/wordclouds, et de la vignette {bib2f} https://cran.r-project.org/web/packages/bib2df/vignettes/bib2df.html
Packages
Pour explorer les entrées bibliographiques, j’utilise les packages {bib2df}, {dplyr}, {tidyr}, {tidyr}, {ggplot2} et {ggthemes}. bib2df} permet de convertir directement les entrées bib en dataframe, ce qui simplifie beaucoup la manipulation des données par la suite. J’utilise {dplyr} et {tidyr} pour la manipulation des données et {ggplot2} pour la visualisation des données.
Pour construire les nuages de mots à partir des titres de mes entrées bibliographiques, j’utilise les packages {tm}, {wordcloud} et {wordcloud2}. {tm} permet d’appliquer des fonctions de transformation à un document texte. Ce package est utile pour supprimer les accents, les signes de ponctuation, les majuscules… {wordcloud} et {wordcloud2} construisent des nuages de mots à partir de documents texte. {wordcloud2} peut utiliser une image binaire comme masque pour créer le nuage de mots. Tous les packages peuvent être installés à partir de CRAN, sauf {wordcloud2}, qui nécessite la version en développement sur GitHub.
library(bib2df)
library(dplyr)
library(tidyr)
library(ggplot2)
library(tm)
library(wordcloud)
# devtools::install_github("lchiffon/wordcloud2")
library(wordcloud2)
library(glue)
library(forcats)Graphiques: thème et palette
Les commandes ci-dessous me permettent de définir un thème et une palette de couleur communs à tous les graphiques.
# Define a personnal theme
custom_plot_theme <- function(...){
theme_bw() +
theme(panel.grid = element_blank(),
axis.line = element_line(size = .7, color = "black"),
axis.text = element_text(size = 11),
axis.title = element_text(size = 12),
legend.text = element_text(size = 11),
legend.title = element_text(size = 12),
legend.key.size = unit(0.4, "cm"),
strip.text.x = element_text(size = 12, colour = "black", angle = 0),
strip.text.y = element_text(size = 12, colour = "black", angle = 90))
}
# Define a palette for graphs
greenpal <- colorRampPalette(brewer.pal(9,"Greens"))Importation du jeu de données bibliographiques
Tout d’abord, utilisons le package {bib2df} pour charger le fichier .bib contenant toutes les références bibliographiques que j’ai rassemblées au cours de mon doctorat. Ce dossier contient plus de références que ce que j’ai cité dans mon manuscrit de thèse. Le fichier .bib utilisé pour l’analyse ci-dessous peut être téléchargé ici.
myBib <- "PhD_Thesis.bib"df_orig <- bib2df(myBib)
head(df_orig)## # A tibble: 6 x 35
## CATEGORY BIBTEXKEY ADDRESS ANNOTE AUTHOR BOOKTITLE CHAPTER CROSSREF
## <chr> <chr> <chr> <chr> <list> <chr> <chr> <chr>
## 1 ARTICLE enugutti_reg… <NA> <NA> <chr [… <NA> <NA> <NA>
## 2 ARTICLE dumais_analy… <NA> <NA> <chr [… <NA> <NA> <NA>
## 3 ARTICLE sawidis_pres… <NA> <NA> <chr [… <NA> <NA> <NA>
## 4 ARTICLE chen_geometr… <NA> <NA> <chr [… <NA> <NA> <NA>
## 5 ARTICLE harrison_com… <NA> <NA> <chr [… <NA> <NA> <NA>
## 6 ARTICLE uyttewaal_me… <NA> <NA> <chr [… <NA> <NA> <NA>
## # ... with 27 more variables: EDITION <chr>, EDITOR <list>,
## # HOWPUBLISHED <chr>, INSTITUTION <chr>, JOURNAL <chr>, KEY <chr>,
## # MONTH <chr>, NOTE <chr>, NUMBER <chr>, ORGANIZATION <chr>,
## # PAGES <chr>, PUBLISHER <chr>, SCHOOL <chr>, SERIES <chr>, TITLE <chr>,
## # TYPE <chr>, VOLUME <chr>, YEAR <dbl>, URL <chr>, URLDATE <chr>,
## # KEYWORDS <chr>, FILE <chr>, SHORTTITLE <chr>, DOI <chr>, ISSN <chr>,
## # ISBN <chr>, LANGUAGE <chr>Nettoyage du jeu de données bibliographiques
En regardant de plus près la colonne JOURNAL, on constate que le nom des revues n’est pas cohérent. Comme cela va biaiser l’analyse des données, il est important de repérer les revues dont le nom existe sous plusieurs formes et d’apporter les corrections nécessaires.
journal_names <- unique(df_orig$JOURNAL)
head(journal_names)## [1] "Proceedings of the National Academy of Sciences"
## [2] "The Plant Journal"
## [3] "Protoplasma"
## [4] "Science"
## [5] "Faraday Discussions"
## [6] "Cell"J’ai trouvé quelques noms de revues écrits de plusieurs façons. Par exemple : “PNAS”, “Proceedings of the National Academy of Sciences” and “Proceedings of the National Academy of Sciences of the United States of America” correspondent à la même revue. Pour nettoyer les données, j’ai regardé les noms des revues un par un et j’ai essayé de repérer autant de synonymes que possible. Pour réduire le nombre de synonymes, j’ai d’abord passé toutes les lettres en minuscule, et supprimé les espaces en trop et certains caractères spéciaux. Idéalement, ce nettoyage des données devrait même se faire en amont sur les données brutes de Zotero, de manière à conserver une bibliographie homogène….
df <- df_orig %>%
# clean names
mutate(JOURNAL = tolower(JOURNAL) %>%
gsub("\\s+", " ", .) %>%
gsub("\\\\", "", .) %>%
gsub("\\&", "and", .)) %>%
# replace synonyms
mutate(JOURNAL = case_when(
JOURNAL == "nature rewiews molecular cell biology" ~ "nature reviews molecular cell biology",
grepl("proceedings of the national academy of sciences", JOURNAL) ~ "pnas",
grepl("proceedings of the national academy of sciences of the united states of america", JOURNAL) ~ "pnas",
grepl("the plant cell online", JOURNAL) ~ "the plant cell",
TRUE ~ JOURNAL
)) Pour vérifier la propreté des données, je pourrais aussi regarder les noms des auteurs, les années, parfois manquantes ou bizarres, et la colonne “BIBTEXKEY”, qui devrait contenir des entrées bibliographiques uniques, à moins d’avoir des doublons. De plus, pour la propreté générale de la base de données Zotero, il peut également être utile de regarder quelles entrées ont peu ou pas de mots-clés. Les mots-clés sont très utiles pour retrouver rapidement les entrées bibliographiques dans Zotero, mais il est facile d’oublier de les remplir.
Exploration du jeu de données bibliographiques
Catégorie
Jetons d’abord un coup d’oeil à toutes les catégories de publications qui se trouvent dans ma bibliographie. Je m’attends à un grand nombre d’articles de recherche, mais je sais que j’ai aussi quelques packages R par exemple.
count(df, CATEGORY, sort = TRUE)| CATEGORY | n |
|---|---|
| ARTICLE | 453 |
| MISC | 9 |
| INCOLLECTION | 4 |
| BOOK | 2 |
| INPROCEEDINGS | 2 |
| PHDTHESIS | 1 |
Sans surprise, la plupart des publications de ma bibliographie de doctorat sont des articles de recherche. Il y a aussi deux livres. Dans ma bibliographie, les publications dans “INCOLLLECTION” correspondent à des sections de livres et celles dans “INPROCEEDINGS” à des articles de conférences. Pour finir, la catégorie “MISC” correspond aux logiciels R, Matlab et GIMP, et aux principaux packages R que j’ai utilisés. Le logiciel d’analyse d’image Fiji est cité en tant qu’article.
Cette exploration des données est l’occasion pour moi de rappeler qu’il est important de citer les logiciels, mais aussi les packages et les plugins utilisés dans les articles scientifiques, en particulier ceux qui sont gratuits et open-source. À l’heure actuelle, les citations sont souvent le seul moyen d’assurer la continuité du financement de ces outils.
filter(df, CATEGORY == "MISC") %>%
mutate(TITLE = gsub("\\{|\\}", "", TITLE)) %>%
select(TITLE)## # A tibble: 9 x 1
## TITLE
## <chr>
## 1 R: A Language and Environment for Statistical Computing
## 2 Matlab
## 3 GNU Image Manipulation Program
## 4 moments: Moments, cumulants, skewness, kurtosis and related tests
## 5 raster: Geographic Data Analysis and Modeling
## 6 FactoMineR: Multivariate Exploratory Data Analysis and Data Mining
## 7 RImageJROI: Read 'ImageJ' Region of Interest (ROI) Files
## 8 snow: Simple Network of Workstations
## 9 rgl: 3D visualization device system (OpenGL)Journal
Je suis curieuse de trouver quelle est la revue la plus courante dans ma bibliographie de thèse de doctorat. Ici, je fais cette exploration pour le plaisir, mais je pourrais utiliser cette information pour mieux savoir où je trouve habituellement mes ressources bibliographiques, peut-être pour renforcer ce biais, en m’abonnant aux newsletters ou aux flux RSS de ces revues (car ils sont ma principale source d’information), ou au contraire, pour choisir de diversifier davantage mes sources. Je vais filtrer les données pour ne garder que celles qui ont un nom de journal, compter combien d’occurrences du nom du journal il y a, et arranger par ordre décroissant pour voir les journaux les plus communs en premier. Voyons quel est le top 10.
top_ten_journals <- df %>%
filter(!is.na(JOURNAL)) %>%
group_by(JOURNAL) %>%
summarize(n = n()) %>%
arrange(desc(n)) %>%
top_n(10, n)
ggplot(top_ten_journals) +
geom_col(aes(fct_reorder(JOURNAL, n), n, fill = n),
colour = "grey30", width = 1) +
labs(x = "", y = "", title = "Top 10 journals in my bibliography") +
coord_flip() +
scale_fill_gradientn("n", colours = greenpal(10)) +
scale_y_continuous(expand = c(0, 0)) +
scale_x_discrete(expand = c(0, 0)) +
custom_plot_theme()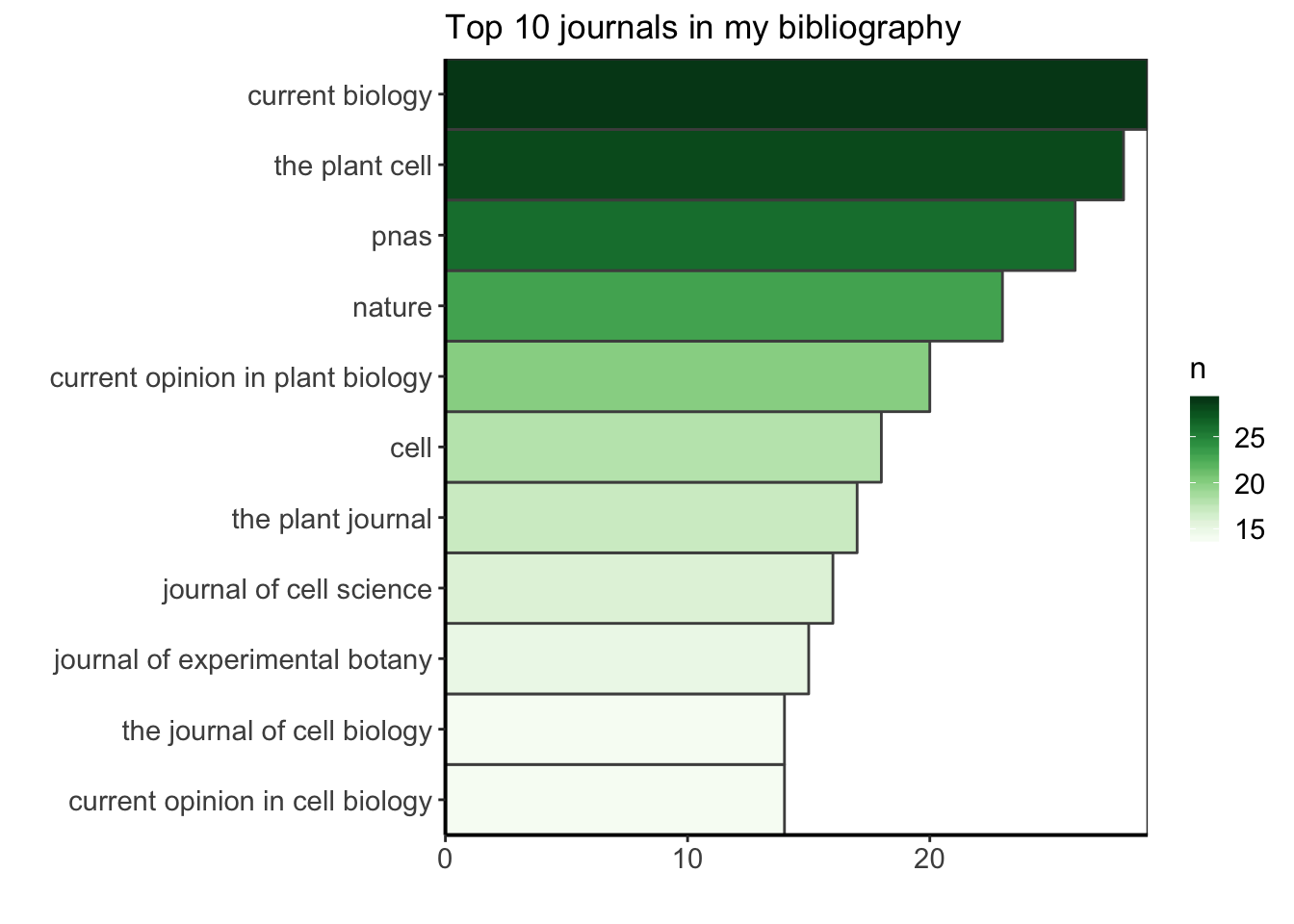
Année
Une autre mesure intéressante est l’année médiane des publications dans ma bibliographie. Ai-je surtout rassemblé des documents récents ? Je vais comparer cette année médiane à l’année où j’ai soumis mon manuscrit et soutenu ma thèse de doctorat (2015) et calculer le temps médian entre 2015 et l’année de publication du document.
year_pub <- df %>%
filter(is.na(YEAR) == FALSE) %>%
mutate(YEAR = as.numeric(YEAR)) %>%
mutate(age = 2015 - YEAR) %>%
summarize(median_age = median(age),
median_year = median(YEAR))
year_pub## # A tibble: 1 x 2
## median_age median_year
## <dbl> <dbl>
## 1 6 2009La moitié des publications avaient 6 ans ou moins au moment de la soumission de mon manuscrit de doctorat, ce qui signifie que j’ai rassemblé principalement des publications récentes dans ma bibliographie.
Regardons maintenant de manière graphique le nombre de publications par année pour voir comment est la distribution, en y ajoutant l’année médiane des publications avec une ligne verticale.
df %>%
filter(is.na(YEAR) == FALSE) %>%
ggplot(aes(x = as.numeric(YEAR))) +
geom_bar(width = 1, fill = greenpal(6)[4]) +
geom_vline(data = year_pub, aes(xintercept = median_year),
colour = "grey30", size = 2) +
labs(x = "", y = "") +
ggtitle("Number of publication per year") +
custom_plot_theme() +
theme(axis.text.x = element_text(angle = 45, hjust = 1))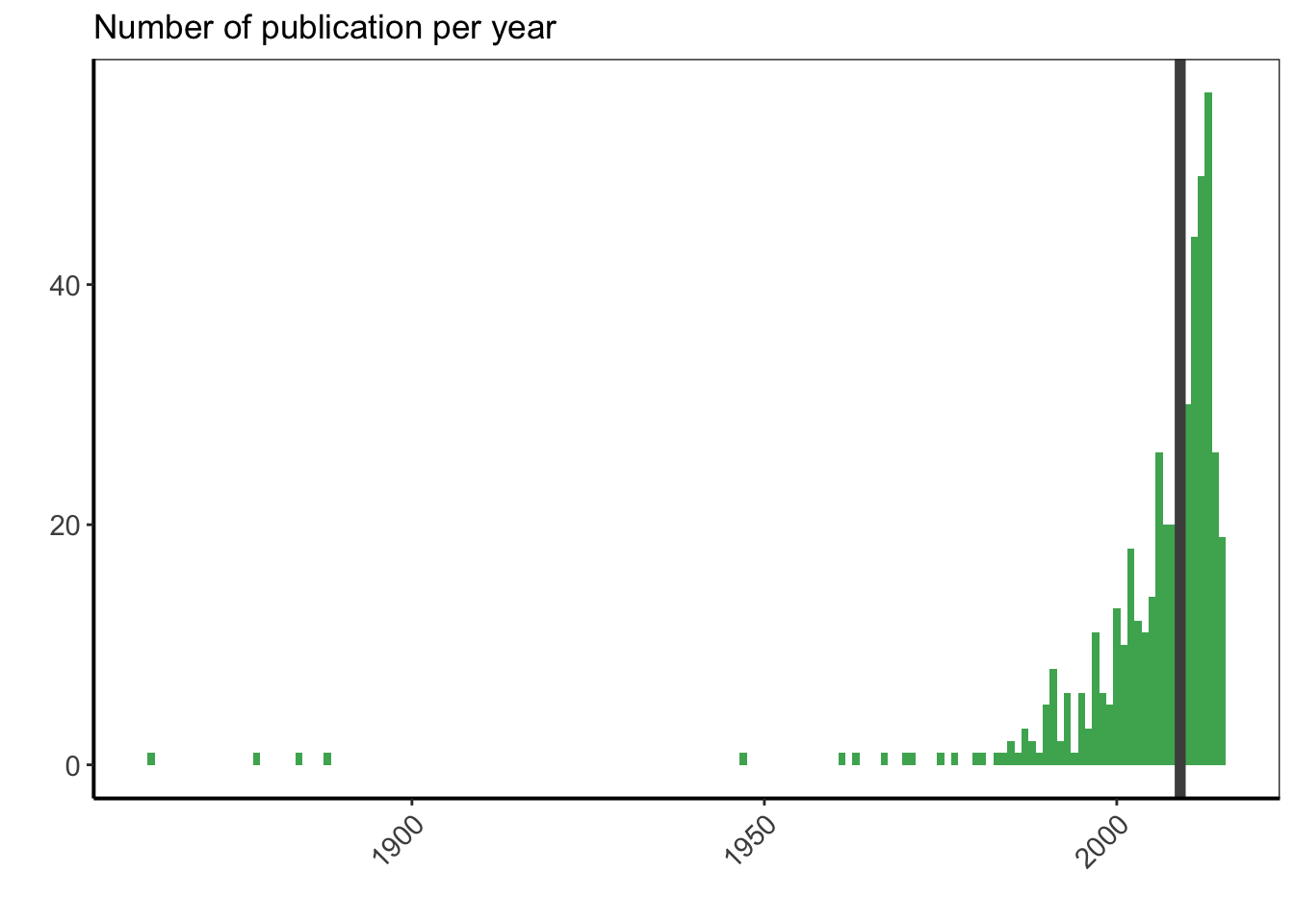
Les publications les plus anciennes correspondent aux règles de division cellulaire du XIXème siècle. Ensuite, j’ai un article de 1947, et toutes les autres publications que j’ai sont d’après 1950. La moitié d’entre elles ont même été publiées en 2009 ou après. Il n’est pas surprenant de voir plus de publications récentes, puisque le nombre de publications par année n’a cessé d’augmenter entre les années 50 et maintenant, et que les publications les plus récentes sont plus facilement accessibles en ligne. Cependant, je suis surprise de voir que l’année médiane n’est que de 6 ans avant la soumission de mon manuscrit de doctorat.
Auteurs
Je suis curieuse de voir quels sont les auteurs les plus fréquents dans ma liste de publications. Les initiales du prénom peuvent plus facilement contenir des fautes de frappe, car elles ont souvent été entrées manuellement. Par ailleurs, il se peut que certaines soient parfois manquantes (dans le cas de prénoms multiples par exemple). Nous commencerons donc par utiliser uniquement le nom de famille.
top_ten <- df %>%
select(AUTHOR) %>%
unnest() %>%
extract(AUTHOR, into = "familyName", regex = "(.*)(?=,)") %>%
mutate(familyName = tolower(familyName)) %>%
count(familyName) %>%
arrange(desc(n)) %>%
top_n(10, n)
top_ten## # A tibble: 10 x 2
## familyName n
## <chr> <int>
## 1 traas 25
## 2 hamant 23
## 3 lloyd 18
## 4 meyerowitz 16
## 5 boudaoud 15
## 6 smith 15
## 7 wasteneys 12
## 8 wang 10
## 9 gibson 9
## 10 li 9Le risque d’utiliser uniquement le nom de famille est que des noms de famille courants comme Smith, Li, Gibson ou Wang soient tous attribués à la même personne. Pour éviter ce problème, je peux ajouter les initiales, en gardant à l’esprit qu’elles peuvent être plus sujettes aux fautes de frappe que le nom de famille lui-même. Pour limiter ce risque, je supprime tous les signes de ponctuation et ne conserve que la première initiale.
top_ten_with_initials <- df %>%
select(AUTHOR) %>%
unnest() %>%
extract(AUTHOR, into = c("familyName", "initials"), regex = "(.*)(?=,)(.*)") %>%
mutate(familyName = tolower(familyName)) %>%
mutate(initials = toupper(initials) %>%
gsub(pattern = "[[:punct:]]", replacement = " ") %>%
gsub(initials, pattern = " ", replacement = "") %>%
substr(1,1)) %>%
group_by(familyName, initials) %>%
count(familyName) %>%
ungroup() %>%
mutate(name =
glue("{substr(toupper(familyName), 1, 1)}",
"{substr(familyName, 2, nchar(familyName))}, {initials}.")) %>%
arrange(desc(n)) %>%
top_n(10, n)
ggplot(top_ten_with_initials) +
geom_col(aes(fct_reorder(name, n), n, fill = n),
colour = "grey30", width = 1) +
labs(x = "", y = "", title = "Top 10 authors in my bibliography") +
coord_flip() +
scale_fill_gradientn("n", colours = greenpal(10)) +
scale_y_continuous(expand = c(0, 0)) +
scale_x_discrete(expand = c(0, 0)) +
theme_classic()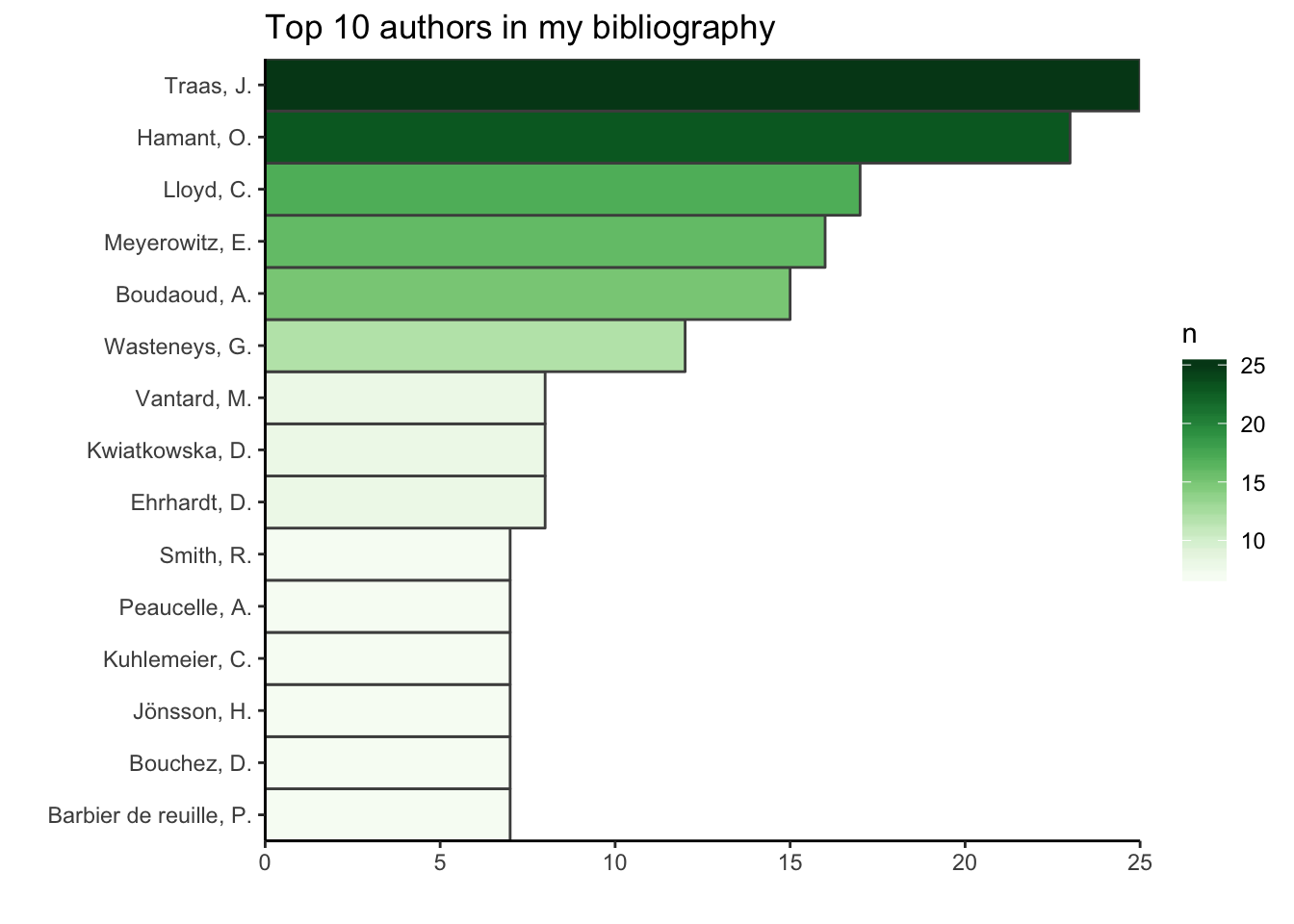
Si l’on ajoute la première initiale, on voit que “Smith” reste dans le top dix mais n’a plus que 7 citations (les 8 autres appartenant à d’autres “Smith” avec d’autres initiales), alors que “Li” ne fait plus partie du top dix. Comme pour les noms de revues, pour avoir des données de bonne qualité, il serait nécessaire de vérifier les noms des auteurs un par un.
Wordcloud : démarrage rapide
Pour visualiser le sujet principal de ma bibliographie, le nuage de mots est l’une des visualisations de données les plus appropriées, car la taille des mots dans le nuage reflète directement leur importance. Je n’utiliserai ici que le titre des publications pour mon corpus.
Pour commencer, je n’utiliserai que les librairies {tm} et {wordcloud}.
pubcorpus <- Corpus(VectorSource(df$TITLE)) %>%
tm_map(content_transformer(tolower)) %>%
tm_map(removePunctuation) %>%
tm_map(removeWords, stopwords('english'))my_wordcloud1 <- wordcloud(pubcorpus, max.words = 200, random.order = FALSE)
Wordcloud : version avancée
Au-dessus, j’ai nettoyé rapidement le corpus en passant toutes les lettres en minuscules et en supprimant les signes de ponctuation. Dans cette deuxième partie, je vais faire un nettoyage plus approfondi du corpus avant de créer le wordcloud. Encore une fois, mon corpus est basé sur les titres des publications de ma bibliographie de doctorat. Je vais réutiliser le package {wordcloud} mais aussi le package {wordcloud2}. {wordcloud2} permet d’utiliser un masque binaire pour créer le nuage de mots.
docs <- SimpleCorpus(VectorSource(df$TITLE),
control = list(language = "en"))Si nous inspectons le corpus, nous constatons immédiatement qu’il a besoin d’être un peu nettoyé : il y a des signes de ponctuation, des majuscules et minuscules, des mots de liaison…
inspect(docs)Transformation du texte
Pour modifier un document texte, par exemple pour remplacer des caractères spéciaux, on peut utiliser la fonction tm_map de la bibliothèque {tm}. tm_map applique une transformation donnée à un corpus et renvoie le corpus modifié. Pour créer nos propres transformations, nous pouvons utiliser la fonction content_transformer du package {tm}. La syntaxe est la suivante (la partie à modifier est en majuscule) :
CHOOSE MEANINGFUL NAME <- content_transformer(
function(x , pattern ) gsub(pattern, CHOOSE REPLACEMENT, x))
)Retirer la ponctuation
toSpace <- content_transformer(
function(x , pattern ) gsub(pattern, " ", x))
docs <- tm_map(docs, toSpace, "[[:punct:]]")Retirer les accents
toNoAccent <- content_transformer(
function(x , pattern ) gsub(pattern, "a", x))
docs <- tm_map(docs, toNoAccent, "à|ä|â")
toNoAccent <- content_transformer(
function(x , pattern ) gsub(pattern, "e", x))
docs <- tm_map(docs, toNoAccent, "è|é|ê|ë")
toNoAccent <- content_transformer(
function(x , pattern ) gsub(pattern, "o", x))
docs <- tm_map(docs, toNoAccent, "ô|ö")
toNoAccent <- content_transformer(
function(x , pattern ) gsub(pattern, "u", x))
docs <- tm_map(docs, toNoAccent, "û|ü")
toNoAccent <- content_transformer(
function(x , pattern ) gsub(pattern, "i", x))
docs <- tm_map(docs, toNoAccent, "î|ï")
toNoAccent <- content_transformer(
function(x , pattern ) gsub(pattern, "oe", x))
docs <- tm_map(docs, toNoAccent, "œ")Passer le texte en minuscule
docs <- tm_map(docs, content_transformer(tolower))Retirer les nombres tous seuls
docs <- tm_map(docs, toSpace, " [[:digit:]]* ")Retirer les mots de liaisons anglais
docs <- tm_map(docs, removeWords, stopwords("english"))Retirer mes propres mots
rm_words <- c("using")
docs <- tm_map(docs, removeWords, rm_words) Pour un corpus non scientifique nous pourrions utiliser le package {pluralize}, mais ici il ne fonctionne pas bien avec les mots scientifiques tels que microtubule/microtubules.
tosingular <- content_transformer(
function(x, pattern, replacement) gsub(pattern, replacement, x)
)
docs <- tm_map(docs, tosingular, "plants", "plant")
docs <- tm_map(docs, tosingular, "microtubules", "microtubule")
docs <- tm_map(docs, tosingular, "cells", "cell")
docs <- tm_map(docs, tosingular, "forces", "force")
docs <- tm_map(docs, tosingular, "walls", "wall")
docs <- tm_map(docs, tosingular, "divisions", "division")
docs <- tm_map(docs, tosingular, "models", "model")
docs <- tm_map(docs, tosingular, "proteins", "protein")
docs <- tm_map(docs, tosingular, "dynamics", "dynamic")Eliminer les espaces en trop
docs <- tm_map(docs, stripWhitespace)Construction d’une matrice term-document
La matrice term-document est un tableau de contingence avec tous les mots et le nombre d’occurence de chacun. Maintenant que le corpus est nettoyé, nous pouvons regarder les dix premiers mots utilisés dans les titres des publications.
dtm <- TermDocumentMatrix(docs)
m <- as.matrix(dtm)
v <- sort(rowSums(m), decreasing = TRUE)
d <- data.frame(word = names(v), freq = v)
d %>%
top_n(10, freq) %>%
ggplot() +
geom_col(aes(fct_reorder(word, freq), freq, fill = freq),
colour = "grey30", width = 1) +
labs(x = "", y = "", title = "Top 10 words") +
coord_flip() +
scale_fill_gradientn("freq", colours = greenpal(10)) +
scale_y_continuous(expand = c(0, 0)) +
scale_x_discrete(expand = c(0, 0)) +
theme_classic()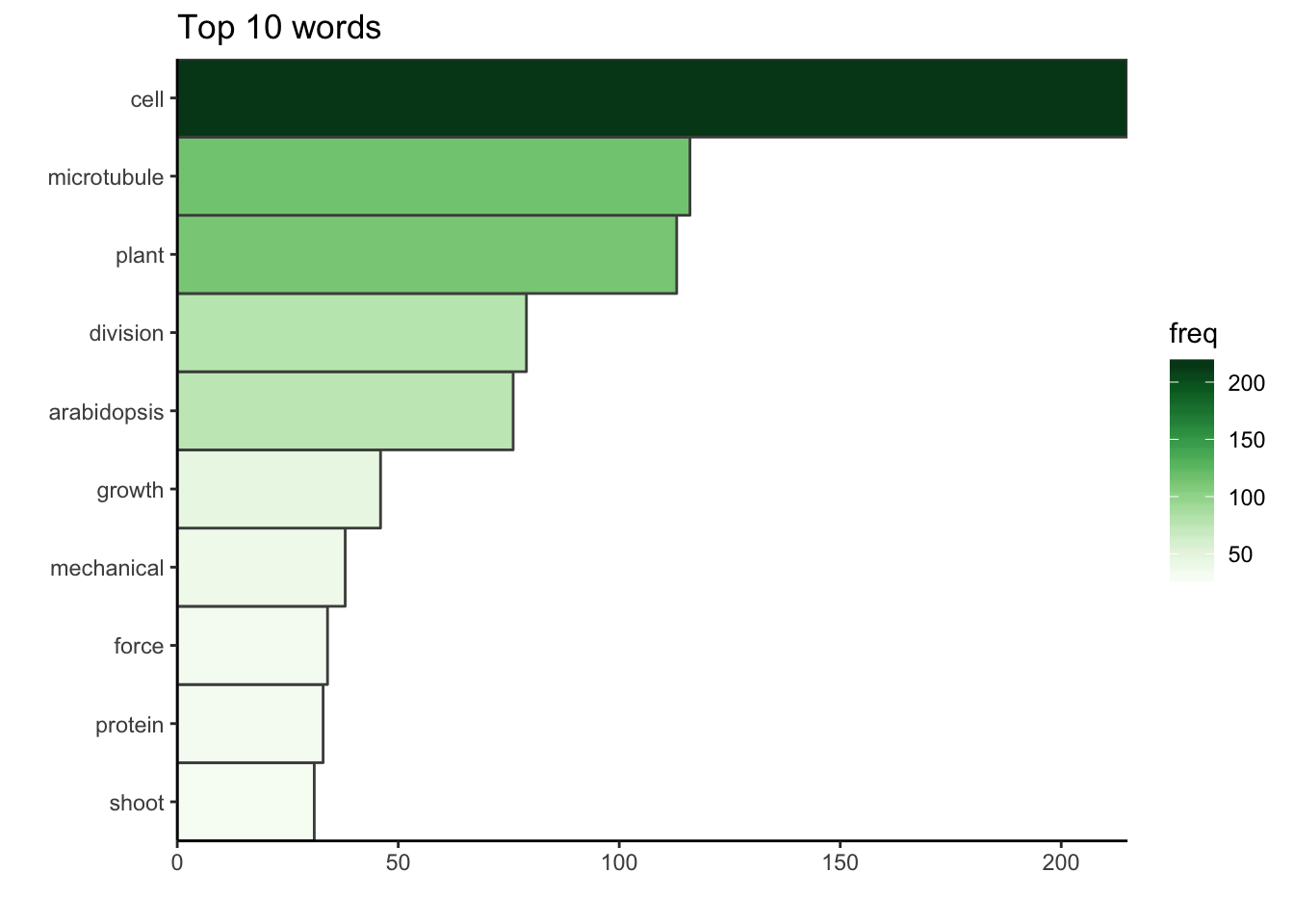
Le mot le plus commun dans le corpus est le mot “cellule”. Pendant mon doctorat, j’ai travaillé sur la division cellulaire chez les plantes, c’est-à-dire comment une cellule végétale partage son volume pour former deux cellules filles. Plus précisément, je cherchais à comprendre dans quelle mesure les contraintes mécaniques au sein d’un tissu végétal en croissance peuvent affecter cette partition en deux cellules filles. Il n’est donc pas surprenant de voir que la bibliographie que j’ai recueillie lors de mon doctorat est principalement centrée sur les cellules (biologiques), mais aussi sur les microtubules, des composants du cytosquelette qui sont des acteurs clés dans la division cellulaire végétale, et bien sûr, sur les plantes, la division (cellulaire) et sur arabidopsis, le nom de la plante modèle que j’ai étudié. Les autres mots-clés décrivent aussi très bien mon doctorat.
Affichage des wordclouds
Commençons par faire un premier nuage de mots simple avec le corpus nettoyé en utilisant le package {wordcloud}. Comparé à celui ci-dessus, il a quelques couleurs, et j’ai utilisé set.seed() pour avoir toujours la même apparence graphique.
set.seed(500)
my_wordcloud <- wordcloud(words = d$word, freq = d$freq, min.freq = 1,
max.words = nrow(d), random.order = FALSE, rot.per = 0.35,
scale = c(6, 0.25),
colors = brewer.pal(8, "Dark2"))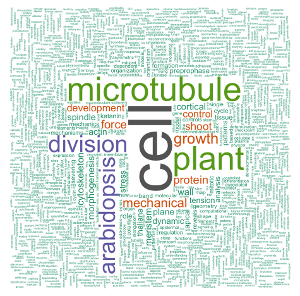
Maintenant, utilisons {wordcloud2} avec une image de fond. J’utiliserai une image de feuille en arrière-plan comme référence à mon sujet de thèse plutôt végétal. Cette image de feuille est une adaptation de cette autre image réalisée par Sébastien Rochette, qui peut être téléchargée ici.
La capture d’écran du nuage de mots html est réalisée manuellement sur mon navigateur web, aux dimensions choisie. J’aurais pu utiliser {webshot}, mais l’utilisation d’une figure comme masque nécessite de rafraîchir le navigateur, ce qui n’est pas facile avec {webshot}.
figPath <- "leaf.png"l <- nrow(d)
# Change to square root to reduce difference between sizes
d_sqrt <- d %>%
mutate(freq = sqrt(freq))
my_graph <- wordcloud2(
d_sqrt,
size = 0.5, # sqrt : 0.4
minSize = 0.1, # sqrt : 0.2
rotateRatio = 0.6,
figPath = figPath,
gridSize = 5, # sqrt : 5
color = c(rev(tail(greenpal(50), 40)), rep("grey", nrow(d_sqrt) - 40)) # gris et vert
# color = c(rev(tail(greenpal(50), 40)), rep(greenpal(9)[3], nrow(d_sqrt) - 40)) # vert
# color = c(rev(tail(greenpal(50), 40)), rep("#FFAC40", nrow(d_sqrt) - 40)) # orange et vert
)Les couleurs choisies pour le wordcloud sont le gris et le vert. Les commandes pour mettre le wordcloud tout en vert, ou bien en orange et vert sont en commentaire.

Conclusion
C’était cool d’explorer cet ensemble de données bibliographiques trois ans après la fin de mon doctorat. Ça a été l’occasion pour moi d’apprendre à jouer avec les données bibliographiques avec R, de mettre en pratique ma connaissance du tidyverse, et d’apprendre à faire des nuages de mots. J’ai découvert quelques trucs auxquels je ne m’attendais pas, comme le fait que l’année médiane de publication dans ma bibliographie n’est que 6 ans avant la soumission de mon manuscrit de thèse et ma soutenance de thèse. J’ai également obtenu quelques perspectives intéressantes sur mon travail et mon profil à l’époque.
Je suppose que les mots clés de ma bibliographie de postdoctorat refléteraient aussi la biologie du développement des plantes, mais aussi très probablement un peu plus la microscopie, en particulier la microscopie à feuille de lumière, et l’analyse de données et des images biologiques. Je travaille aujourd’hui encore plus avec des logiciels d’analyse d’images comme MorphographX et ImageJ/Fiji. Et je travaille encore plus avec le logiciel R, avec lequel j’étends mes workflows d’analyse de données issues d’images biologiques à travers des packages R que je développe comme {mgx2r}, {mamut2r} ou {cellviz3d}.
Remerciement
J’aimerais remercier le Dr. Sébastien Rochette pour son aide pour nettoyer le corpus avant de générer les nuages de mots.
Citation :
Merci de citer ce travail avec :
Louveaux M. (2018, Dec. 21). "Analyse de références bibliographiques avec R". Retrieved from https://marionlouveaux.fr/fr/blog/bibliography-analysis/.
@misc{Louve2018Analy,
author = {Louveaux M},
title = {Analyse de références bibliographiques avec R},
url = {https://marionlouveaux.fr/fr/blog/bibliography-analysis/},
year = {2018}
}


Partager ce post
Twitter
Google+
Facebook
LinkedIn
Email